文:回溯档案
编辑:回溯档案
棉花(棉特别方案,主要是四倍体,棉布L.,产于世界上许多不同的热带、亚热带和暖温带地区,因为棉花种植如此广泛,中国2016年,全球面积为3,100万公顷的棉花从植物繁殖中获得的社会效益大大扩大。

这些措施包括改善自然资源的保护和保存,减少对不受欢迎的化学品保护剂的依赖,提高生产的时间和能源效率,增加种植者和与棉花有关的企业以及地方甚至国家经济的利润。
通过育种棉质资源,特别是遗传优良品种,基本作物产量和产品质量有可能得到改善,由于分子标记和序列多态性促进了种质分析、分类、分类、基因型化、基因组比较,以及各种标记辅助选育用于育种和繁殖相关研究。
棉花纤维作为一种商品的重要性因其在纺织品中的广泛使用而得到广泛承认,棉籽作为食用油和牛饲料来源的重要性很少得到承认,也很少得到研究。
对棉籽的新兴趣,以供非反胃动物甚至人类食用,是因为越来越需要负担得起的蛋白质来源来养活日益增长的全球人口以及选择性清除种子中棉酚的能力,饲料和食品需求的增加可能会随着时间的推移而改变,并增加种子的使用。

无论如何,全球气候变化将要求品种适应较高的温度、降水量减少和/或盐度增加,以及新的基因型,对改变的害虫和病原体分布具有抵抗力。为了应对这些挑战,植物育种者需要找到新的变异来源,并将其纳入育种计划。

世界范围内其他棉花种质收藏中的各种资源也已通过分子标记,主要是SSRS来确定特征,此外选育计划中的优秀品种往往具有基因型,以便能够对特定国家和生长地区的品种、已知家系和各自选育的种质资源进行分子比较。

如果将有关遗传差异的知识积累与有关表型差异的知识结合起来,则尤其有用,因为它使育种者和其他研究人员能够通过选择具有互补表型特征和遗传差异性的父母,产生更加有用的分离群体。
然后利用这些种群组合并选择合适的等位基因,绘制并确定特定表型的遗传基础;启动先进品种的开发。

随着可用于种质保存的资源不断减少,优化这些资源变得至关重要,未经确认的收藏中的种质冗余在阻碍最佳管理和利用收藏方面发挥了重大作用。
在水稻种质库中,只有三分之一的加入可能是不同的,这种情况或多或少在许多种质收集中得到承认,任何植物收集的管理都需要大量的资源来维持一个高度可行的种子库存,以确保种子随时可以从收集中得到利用。

对于棉花,许多生产线的光周期敏感性要求在两个或更多纬度地区进行昂贵的田间和苗圃生产,因此重复材料的维护导致大量不必要的努力和费用。
管理种质收集和育种方案的另一个关切是保持纯度,过交会损害线条的纯度,其中大部分不能很容易地通过表型观察来检测,在商业品种纯度测试中,除表型外,还越来越多地依赖准确的基因型谱。
基因型特征分析是一种寻求于种质资源管理的工具,与棉花的种质集合有关,棉花是自花授粉的,但非常适合昆虫诱导的异花授粉。
最近在棉花中,SSRS一直是研究人员识别基因型差异的首选标记,一般而言,r标记可以检测到多个等位基因,从而产生高多态性,从而能够利用较少数量的标记。

然而,使用SSRS进行基因分型耗时、成本适中,而且在高吞吐量的情况下很难做到,相比之下,单核苷酸多态性(SNPS)在每个基因点上只有两个等位基因,因此在一个基因点上单个标记的信息量要比在一个rps上的信息量小。
然而,在整个基因组中,SNPS比SSRS发生得更频繁,可以获得更多的标记。SNPS的好处是,由于双等位基因的存在,可以用高吞吐量、成本效益高的方式同时对大量SNPS进行基因型。

当用相同数量的玉米SSRS和SNPS进行多样性分析时,SSRS分析通常信息量更大,但当SNPS的数量增加时,这种限制就被克服了,SNPS能够解决极为相似的个人之间的差异,并提高多样性估计的准确性。
为了更透彻地描述棉花的遗传多样性,我们利用Cottonsnp63K阵列分析了由全国棉花理事会和世界各地的合作者提供的395个棉花样本,抽样收集的数据表明,这项技术如何应用于评估遗传多样性和群体间的种质结构。

植物材料和基因型
改进的G.hirsutum被选为跨全球棉花育种工作区,在改良型中,美国开发了185个品种,这些品种又进一步分为四个繁殖区,东部、中南部、平原和西部。
关于用于分类到全球和美国繁殖区的标准的详细说明,分类依据的是提交时申报的护照数据,以延续我们先前的报告,非洲的基因型分为两组,北非和南部非洲,赤道为边界。

我们的理论认为,非洲改进后的种质之间的遗传差异将较少基于地理空间考虑,而更多地基于繁殖史,有人认为,基因群可能存在于以前的殖民和贸易关系基础上。
这两个区域集团是武断的,但为了保持连续性而予以保留,在改进的小组内G.hirsutum其中包括15个名称相同的基因型的例子,以检查名称相同的基因型是否也具有相似的基因。
在12个例子中,基因型来自不同的育种程序(生物复制),在其余例子中,基因型来自同一种子来源.
根据胡尔斯-肯普的研究结果,为每个小组成员生成了SPP基因型,使用科顿斯佩63K阵列,其中包含63,058个SNP英菲尼铵II测试和四倍体的集群文件。

集群文件的开发使用了1,156个棉花样本,包括在这个多样性分析中使用的植物材料的子集,在本分析中,所有样本的基因型都是使用Hules-肯普分类为多态性的38,822SNPs确定的。
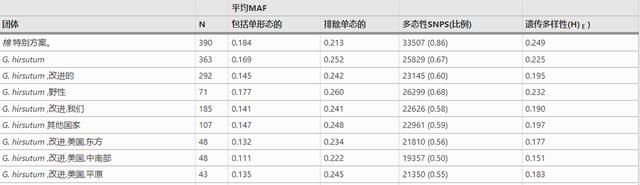
小等位基因频率 棉 种质组
我们利用高通量基因型平台来评价栽培棉花及其野生亲缘关系的多样性,在本研究中确定的SNPS中,33,507个(86%)是多态的。

从20,000个序列中选择来检测G.hirsutum 以及其他五种物种,该阵列能够检测到17,934个种间多态性(种间SNPS),这对于内插研究和繁殖是非常有用的。

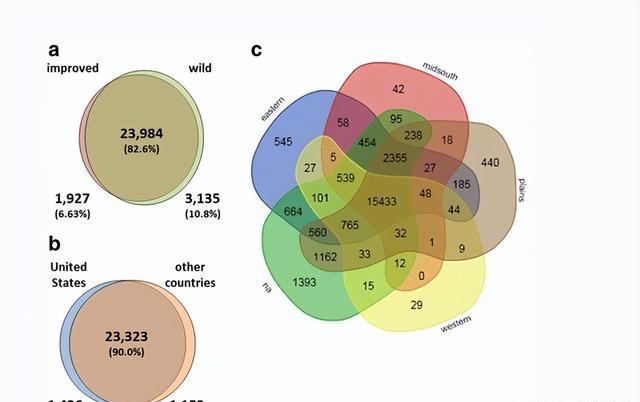
最初的特点是,在几个群体中,它们是独一无二的,在非改良型和改良型的比较中,大部分SNPS(23984;82.6%)是两个群体共同的,表明大多数野生型的遗传变异也在改良型中发现。
遗传相似性与遗传关系 棉 繁殖群体
用两对个体之间相同的状态值(IBS)来估计特定育种组内的平均遗传相似性。两个组别的G.hirsutum 不同品种之间的相似性最高(IBS=0.704),野生类型之间的相似性最低(IBS=0.652)。

来自美国的品种比来自其他国家的品种稍相似(IBS=0.683),在美国育种区内,中南方品种之间的相似性最高(IBS=0.739),而最不相似的品种起源于平原地区。
遗传相同系的表型评价
在被确定为高度相似的入会对的表型比较中(IBS&ttt;0.98),26个形态描述符中只有7个在入会对中存在差异。叶和铃蜜腺分布的描述符评级在这些配对的品种中最为常见,其次是茎毛数量的评级差异。

尽管这对组合有着完全的遗传相似性(IBS=1000),但一对组合(md51ne和siokra104-90)在五个描述项上有所不同,没有共同的繁殖史支持相同的基因型这两个群体之间。
用于基因鉴别和表型分析的植物来自于从国家遗传控制中心获得的种子,但每次评价都使用不同的植物,因此在取样和/或处理方面有可能出现错误,无意中可能导致了这些观察。
棉花中SNP标记与SNP标记的比较
下一代测序技术的发展以及由此产生的数千个标记(主要是SNPS)的检测,提高了分析种质收集多样性的能力,以及利用这种多样性来推动植物育种计划。

我们调查了棉 利用最近开发的Cottonsnp63K阵列和390个大型面板的物种棉 样品包括292个改良品种,G.hirsutum 以及少量未经改进的G.hirsutum 其他棉 物种。
这一数据使我们得以评价该阵列对评价遗传多样性和种群结构的效用,并对美国农业部国家棉花种质收集的很大一部分进行基于SNP的定性和分析。
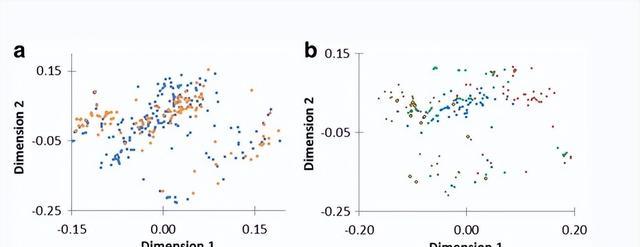
所观察到的棉 物种和内部G.hirsutum 同意根据以往的研究和分子标记而作出的预期,标记物清晰地分开G.hirsutum 把野生动物与其他物种区别开来G.hirsutum .
在改进的G.hirsutum ,如其他SNP研究所述,已观察到的聚类可能显示轻微的判断偏倚,样品类似于MTM-1的样品被定位在两个改进组的底部G.hirsutum .我们发现该阵列可以有效地利用PCOA和结构分析对种质进行分类。

结论
随着SNP发现项目的不断增加,以及COttonsnp63K阵列的发展,可以预测SNP标记在棉花遗传学、种质保护和育种应用中将发挥越来越重要的作用。
在本研究中,我们提供了一个大型的全基因变异数据集,主要种植棉花,在有代表性的棉花基因型中,成千上万的SNPS提供了在世界各地种植的棉花之间进行精细鉴别的机会。
SNPS将作为紧密的基因组变异标记,用于相关映射方法,目的是将分子多态性与表型性状的变异联系起来,以及用于棉花的分子育种方法。
参考文献
【1】阿尔福德,顾立帕,凡贝。棉籽蛋白:未来会是什么?植物食品嗡嗡声。1996年;49:1-11。
【2】罗托雷,苏尼勒库马尔。美国专利申请,2013,314572。申请日期。2011年。
【3】欣策,方德,戈尔M,谢弗勒B,尤杰,弗赖利乔夫斯基,珀西R。的分子特性棉 美国全国棉质资源多样性参考集。神学院。2015年;128:313-27。
【4】布鲁贝克公司,文德尔律师事务所。重新评估驯化棉花的原产地(棉布 使用核限制片段长度多态性(RFPS)。我是机器人。1994年;81:1309-26。
【5】卢贝尔斯,奇普。全球基因库G.hirsutum 以及它的改进。在:帕特森啊,编辑。棉花的遗传学和基因组学。纽约:斯普林格;2009年。P.23-52.
