转自:生物谷
文章解读+创新点拓展,为您带来科研新体验~
导读
在生物医学研究中,细胞特异性反式调控元件(cell-type-specificcis-regulatoryelement,CRE)的设计是一个热门研究领域。最近发表于《Nature》的一项研究《Machine-guideddesignofcell-type-targetingcis-regulatoryelements》,利用机器学习模型指导合成具有高度细胞类型特异性的CRE,为精准医疗提供了全新的工具。这项技术不仅有望解决现有基因治疗中靶向递送不足的问题,还可能为基因编辑、报告基因开发等提供新的解决方案。
研究背景
基因表达是一个复杂的过程,受到多种因素的调控,其中CRE扮演着关键角色。这些DNA序列通过与特定转录因子结合来调节目标基因的活性。尽管近年来的研究已经鉴定了数百万个潜在的人类CRE,但这些自然进化的序列仅仅是所有可能性的一小部分,并不一定符合临床应用中的最佳表达需求。理论上,一段200碱基对的DNA可以包含超过2.58×10120种不同的组合——比可观测宇宙中的原子数量还要多。这些DNA序列组成了一个尚未被充分发掘的DNA序列空间,也是挖掘具有临床和生物技术应用潜力的CRE的储备库。
研究设计与结果
为了克服传统方法在识别和验证CRE时遇到的挑战,Gosai团队引入了三项关键技术:
1)大规模平行报告基因检测(MassivelyParallelReporterAssay,MPRA):能够同时评估成千上万个CRE的功能,从而获得关于调节语法(regulatorygrammar)的广泛认识。
2)深度学习算法(Malinois):用于预测不同细胞类型中遗传序列如何影响CRE活性,构建出精确的“调控语法”模型。
3)基于预测模型的定向CRE生成(CODA):基于上述CRE序列评分算法,对随机序列进行迭代,允许研究人员根据所需功能和特定细胞定制合成CRE。
实验结果显示,使用这些合成的CRE可以在斑马鱼和小鼠中实现细胞类型特异性的驱动效能。此外,该研究也展示了如何利用CODA软件库及Malinois模型来生产合成序列,并通过体外和体内实验验证了其效能。
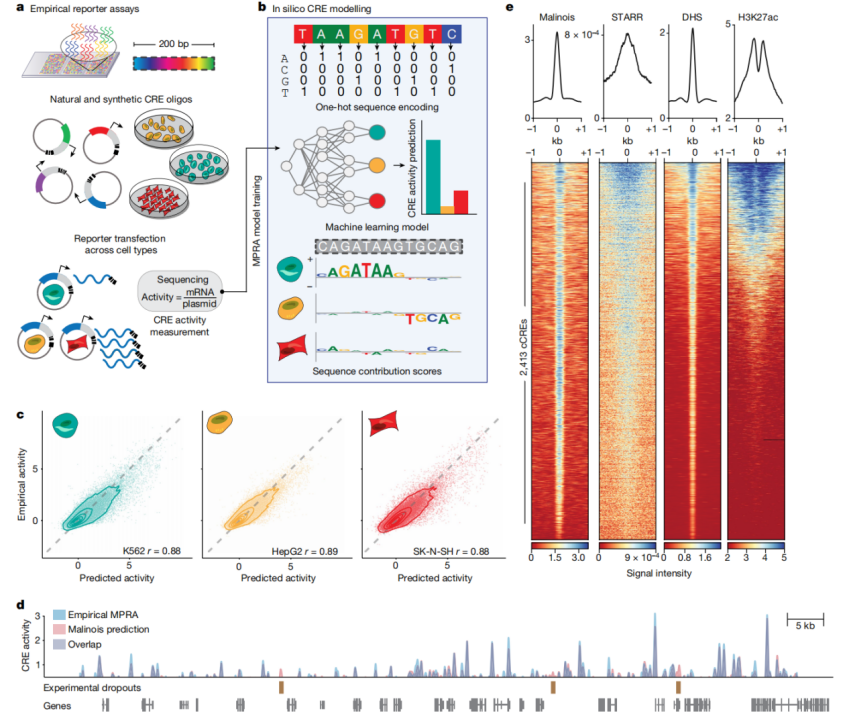
为了建立精确的CRE活性模型,研究人员首先在K562(红系前体细胞)、HepG2(肝细胞)和SK-N-SH(神经母细胞瘤)三种人类细胞类型中通过MPRA分别分析了超过77万段200nt长的DNA序列,筛选具有细胞特异性CRE活性的基因组序列信息(图1a)。
使用这些数据,研究人员借鉴了Basset模型(一个用于预测染色质可及性的模型),构建了基于卷积神经网络(ConvolutionalNeuralNetwork,CNN)的深度学习模型Malonis(图1b)用以预测DNA序列的细胞特异性CRE活性。Malonis在K562、HepG2和SK-N-SH细胞中的预测值与实际测量值之间显示出高度相关性(Pearson’sr=0.88–0.89;Spearman’sρ=0.81−0.83;P
